Mediante programas informáticos de simulación computacional se obtuvo en tercera dimensión (3D) la estructura de una combinación de insulina y IGF1R, proteínas o receptores de la membrana celular que interactúan con la insulina glargina, fármaco por excelencia en el tratamiento de la diabetes mellitus tipo I.
El resultado es un paso importante para entender por qué durante el tratamiento se activan determinadas vías de señalización celular (capacidad de una célula de recibir, procesar o transmitir señales en su entorno y consigo misma), y si en periodos largos el mayor crecimiento de algunos tejidos del cuerpo puede producir otras enfermedades.
La insulina es una hormona producida por el páncreas, crucial para el funcionamiento del organismo humano pues asegura que las células tengan energía y puedan llevar a cabo sus tareas, aunque su función primordial es regular y controlar los niveles de glucosa en sangre previniendo desbalances que dan lugar a enfermedades como la diabetes tipo I, II o gestacional.
Según la Organización Panamericana de la Salud, en América unos 62 millones de personas padecen la enfermedad, especialmente en países de ingresos medios o bajos, y cada año se producen más de 244.000 muertes por esta condición. Por ello, científicos e investigadores de todo el mundo avanzan en la búsqueda de fármacos que ofrezcan mayor fiabilidad en el tratamiento contra la diabetes.
Sin embargo aún existen factores asociados con su desarrollo celular que no se han esclarecido, como por ejemplo cómo es la estructura tridimensional de los receptores híbridos, o sea la combinación entre el receptor de insulina y el 1GF1R, y la forma en que compuestos análogos o similares a la insulina glargina (fármaco por excelencia en el tratamiento de diabetes tipo I) interactúan con estos.
Como una manera de generar mejores tratamientos a futuro, el investigador Luis Fernando Arévalo Corredor, magíster en Ciencias Farmacéuticas de la Universidad Nacional de Colombia (UNAL), simuló, de manera hipotética y por computador, el modelo de este receptor hibrido, lo cual no se había logrado antes con técnicas convencionales como la difracción de rayos x o la criomicroscopia electrónica, pues se tenían dificultades en el laboratorio para evitar las interferencias o restricciones propias del tejido o membrana celular.
Esta técnica utiliza softwares avanzados que emergen como una alternativa para mejorar los tiempos de diseño y síntesis de los fármacos, disminuyendo los costos asociados con experimentos fallidos o poco precisos que no tienen en cuenta las interacciones que se producen entre el receptor y los compuestos.
Según el magíster, los receptores tienen 4 sitios de unión, que son lugares a los que pueden llegar la insulina o sus análogos e interactuar, en este caso el fármaco simulado para la glargina. Se registró que este compuesto tiene una mayor afinidad, o sea que prefiere llegar al primer sitio de unión del receptor, lo que quiere decir que allí es donde se producen la mayor cantidad de cambios y activaciones de vías de señalización celular durante el tratamiento de la diabetes.
Entre los programas informáticos especializados que usó se encuentran Swiss-Model y Prodigy, que simulan estructuras de las proteínas en 3D determinando variables como la constante de afinidad y cómo se dan las interacciones mencionadas.
Gracias a ellos construyó una serie de modelos para observar lo que ocurría en la membrana celular cuando los receptores estaban en presencia de la glargina, y 2 de ellos resultaron idóneos para dilucidar estos procesos, algo que no se había obtenido antes y que supone un paso prometedor para entender la enfermedad y cómo estas interacciones celulares pueden transformar el tratamiento, para bien o para mal.
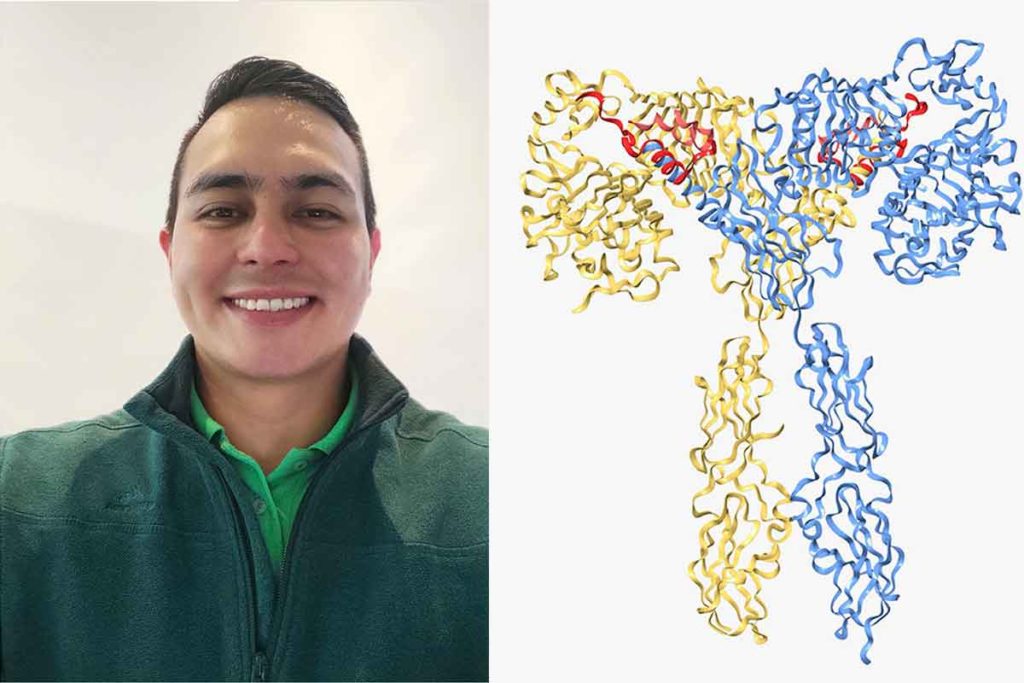
El investigador asegura que “estos modelos hicieron más sencillo el proceso, pues permitieron cargar una gran cantidad de datos de los aminoácidos en cada estructura, y se pudo implementar un molde computacional en el que las interacciones se dilucidaron mejor. Se identificaron hasta 9 interacciones que no se tenían registradas para el análogo de la insulina glargina.
“Son hallazgos fundamentales a futuro, pues, además de acercarse a lo que se puede obtener experimentalmente, ayudan a entender cómo se dan las interacciones y permitirían crear insulina glargina modificada o un nuevo medicamento que no genere problemas como el crecimiento desmedido de algunas células durante el proceso”, explica el experto en Ciencias Farmacéuticas, quien contó con el apoyo y los aportes del Grupo Unimol, del Departamento de Farmacia de la UNAL, liderado por el profesor Claudio Jaime Gómez Alegría.