Un trabajo fin de máster de la UNED ha permitido desarrollar un sistema de inteligencia artificial basado en redes neuronales que identifica el talón de aquiles de las bacterias, es decir, los genes responsables de que muestren resistencia a los atibióticos.

Todo indica que en los próximos años se va a multiplicar el número de personas en el espacio. Por lo que, además de contar con unos medios tecnológicos para sacarlas con seguridad fuera de nuestra atmósfera, con destinos diversos como la Luna, la Estación Espacial Internacional o, en un futuro algo más lejano, Marte, también se debe garantizar la salud en un ambiente tan hostil como el del espacio.
Actualmente se conoce que las estancias prolongadas en la Estación Espacial Internacional tiene consecuencias sobre el sistema inmune, lo que hace a los astronautas especialmente vulnerables, más si se tiene en cuenta que una vez en el espacio, las posibilidades de regresar a la Tierra en el tiempo adecuado para se tratado de una enfermedad son muy escasas o prácticamente nulas.
Por eso, las agencias espaciales están invirtiendo mucho tiempo de investigación en reducir las posibilidades de verse afectado por una enfermedad durante la estancia fuera de la Tierra. Una de sus prioridades es reducir la resistencia bacteriana, es decir, la capacidad de las bacterias para defenderse de los antibióticos.
Qué ha aportado la UNED a la investigación sobre la resistencia bacteriana en el espacio
En este ámbito, el trabajo fin de máster realizado en la UNED por Pedro Madrigal, que actualmente es el investigador del Instituto Europeo de Bioinformática (EMBL-EBI), ha supuesto un salto de calidad muy importante, porque en él se describe una fórmula basada en inteligencia artificial y aprendizaje profundo, que mejora la identificación de genes de la microbioma de la Estación Espacial Internacional.
“La Estación Espacial Internacional tiene una distribución de microorganismos, o microbioma, en sus superficies que es característica de este hábitat pero esto no es una sorpresa. Por ejemplo, utilizando metagenómica para obtener secuencias del genoma de los diferentes microorganismos en estaciones de metro de todo el mundo se han descubierto diferencias de igual magnitud”, explica Pedro Madrigal. Pero hay que conocerla y estudiarla bien.
El objetivo de esta investigación gestada en el Máster en Investigación en Inteligencia Artificial de la UNED ha sido entender cómo afectan la diversidad microbiana y su daño a los vuelos espaciales, así como ayudar a los investigadores y astronautas a abordar el riesgo de infección y resistencia a antibióticos.
Y para ello se han empleado algoritmos de aprendizaje automático, que es la rama de la inteligencia artificial dedicada a automatizar parte del método científico, en el cual las computadoras observan datos para construir un modelo basado en ellos y utilizar a la vez este modelo como una hipótesis a validar, que aporte solución a problemas complejos.
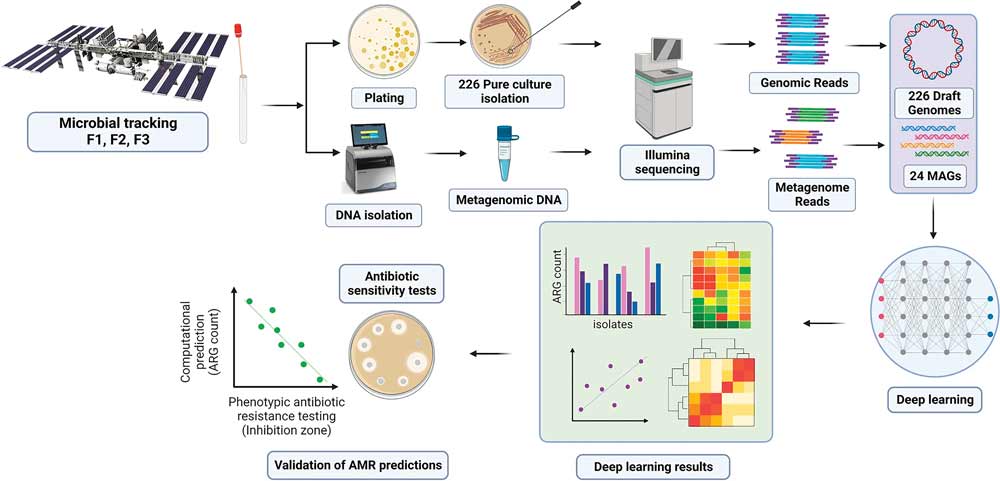
Cómo se descifra la resistencia genética de las bacterias
Las técnicas de secuenciación actuales permiten el acceso directo y la creación de perfiles del grupo total de ADN metagenómico, donde los genes responsables de la resistencia a los antibióticos (conocidos como ARG) generalmente se identifican o predicen en función de los «mejores resultados» de las búsquedas de secuencias en las bases de datos existentes.
Desafortunadamente, este enfoque produce una alta tasa de falsos negativos (genes identificados como no resistentes que sí lo son). Para abordar las limitaciones de esta orientación, se ha utilizado un enfoque de aprendizaje profundo supervisado (Deep learning), en concreto DeepARG desarrollado por otros investigadores en 2018.
Se utilizaron en concreto, los modelos de aprendizaje profundo DeepARG-SS y DeepARG-LS, para secuencias de lectura corta y secuencias de longitud completa del gen, respectivamente y que han demostrado una alta sensibilidad para la detección de genes de resistencia a los antibióticos. Se trata de una red neuronal profunda (deep learning) que considera una matriz de disimilitud entre las diferentes categorías de genes ARG. Esta matriz de disimilitud representa la diferencia existente entre dos determinados ARGs. La capa de salida de la red neuronal en DeepARG está compuesta por 30 unidades que se corresponden con las diferentes categorías de resistencia a antibióticos.
Cómo se han hecho los experimentos
Los resultados del último vuelo revelaron el dominio genes resistentes a antibióticos de Kalamiella piersonii, una bacteria relacionada con la infección del tracto urinario en humanos. En el análisis se emplearon 226 cepas puras aisladas del proyecto MT-1 se detectaron cientos de genes de resistencia a antibióticos, incluidas dos especies de alto rango que correspondían a cepas de Enterobacter bugandensis y Bacillus cereus.
Las predicciones computacionales fueron validadas experimentalmente por perfiles de resistencia a antibióticos en estas dos especies, y se demostró un alto grado de concordancia entre ambas técnicas. Específicamente, los datos del ensayo de disco que confirmaron la alta resistencia de estos dos patógenos a varios antibióticos betalactámicos, que son el grupo de antibióticos más comunes.

Los profesores del departamento de Inteligencia Artificial de la UNED, Elena Gaudioso y Félix Hernández del Olmo, junto con Afshin Beheshti (investigador en NASA Ames Research Center), se han encargado de la dirección de este trabajo, supervisado la formulación de los algoritmos de Deep learning (un tipo de particular de Machine Learning) que se han usado, su aplicación a los datos proporcionados por las instituciones integrantes del proyecto y la validación de los resultados obtenidos.
El siguiente paso, explica Pedro Madrigal, no pasa necesariamente por diseñar nuevos fármacos, aunque sí es cierto que hay compañías tratando facilitar esto mediante la microgravedad. “Las siguientes cuestiones de importancia para las agencias espaciales con respecto a la resistencia antimicrobiana son, por un lado, conseguir un sistema que permita estudiar y monitorizar la evolución de la resistencia antimicrobiana durante vuelos tripulados de larga duración, si es posible en tiempo real”. Por otro lado, el “diseño óptimo de una astrofarmacia para estas misiones”, donde los antibióticos a bordo deben cubrir una amplia gama de potenciales amenazas para la tripulación.
Se ha comprobado que la radiación del espacio incrementa las mutaciones de las bacterias, que acaban generando resistencia a los antibióticos.
Y si eso se une a que el sistema inmune de los astronautas se altera durante el vuelo espacial, se tiene “la tormenta perfecta”. Ahí la importancia de este estudio y otros en la misma línea para conocer mucho mejor a las bacterias.
















