Primero fue China, y se pensaba que era un problema local. Sin embargo, la globalización ha sacado su lado oscuro y ha contribuido a difundir el corona virus por todo el mundo. A pesar de todo, resulta muy difícil seguir el corona virus y se necesitan herramientas basadas en supercomputación para seguir la evolución de la epidemia.
La Universidad de Almería colabora con un grupo de la Complutense de Madrid, en el desarrollo de algoritmos para el seguimiento de las epidemias.
Supercomputación para seguir la epidemia
La aplicación se puso en funcionamiento con la pasada epidemia de ébola del mes de julio y que obligó a poner en marcha todas las alerta en República Democrática del Congo.
Las universidades de Almería y Complutense de Madrid sí han seguido muy de cerca esta enfermedad, con una herramienta especial que, con una simulación, predice cómo se comportará la epidemia.
Ambas universidades colaboran en un proyecto conjunto para el modelado y seguimiento de epidemias, para lo que emplean superordenadores con una capacidad de cálculo enorme, que permiten simular el avance de la enfermedad, una información de mucho interés para el desarrollo de una actuación efectiva para frenar la epidemia.
La colaboración se remonta a unos años atrás, cuando el Instituto de Matemática Interdisciplinar de la Complutense se puso en contacto con el grupo de investigación de Supercomputación y Algoritmos, que trabaja en la Escuela Superior de Ingeniería de la Universidad de Almería.
Buscaban un equipo especializado en la realización de cálculos de gran volumen con superordenadores, y recurrieron a los investigadores almerienses, por su trayectoria en supercomputación y desarrollo de algoritmos específicos para este tipo de operaciones, que a veces llegan a ocupar días enteros de cálculo.
Be-CoDiS, para el ébola y el corona virus
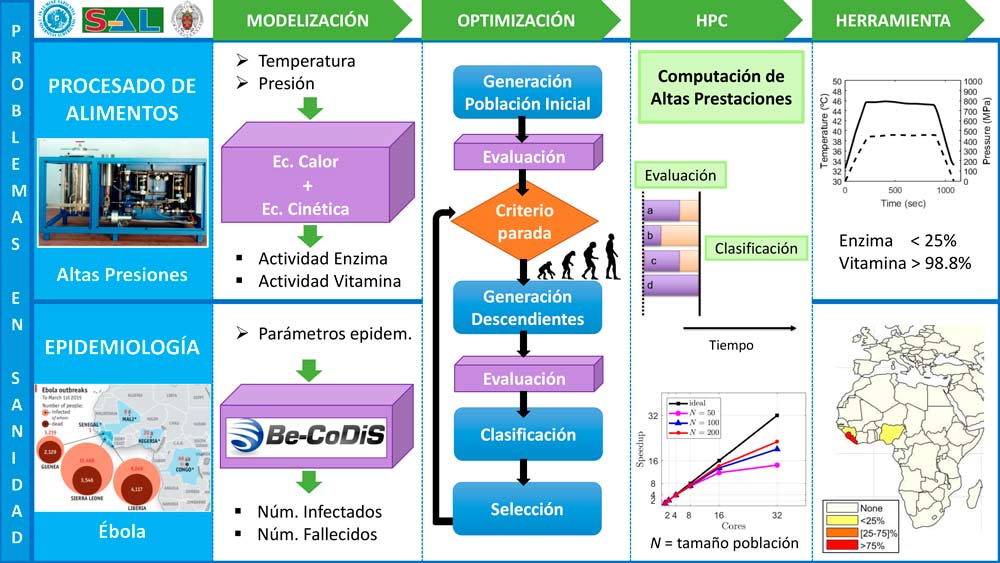
Este órgano de la Complutense desarrolló en 2014 la herramienta Be-CoDiS, para predecir el riesgo de propagación del ébola y que se basaba en el cálculo matemático.
Su problema residía en la enorme capacidad de computación requerida, para simular el avance de la enfermedad en una región determinada, debido a la gran cantidad de variables y de datos que entran en juego, a la hora de desarrollar una imagen del movimiento de la enfermedad con este modelo matemático.
En él se tienen en cuenta factores tan determinantes para la expansión de la epidemia como los flujos migratorios, los efectos de las medidas de control y coeficientes temporales. Datos cruciales para obtener una simulación lo más ajustada a la realidad, pero que requieren una capacidad de cálculo fuera de lo normal.
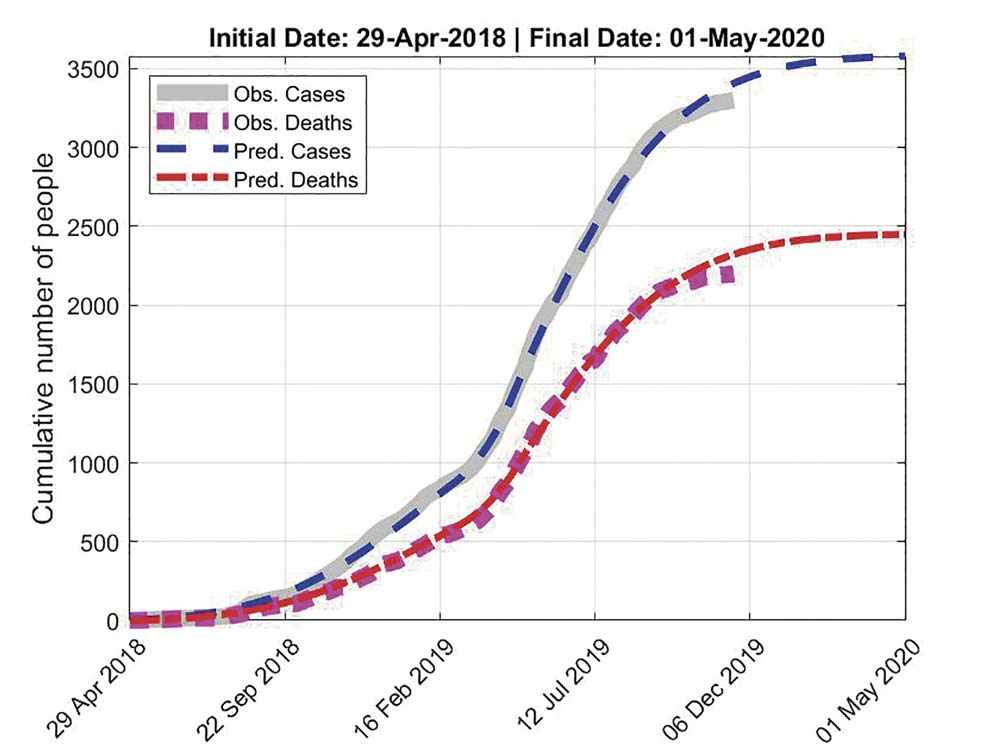
Así, tras realizar el último ajuste del modelo el pasado mes de julio, con los datos de la epidemia de ébola que actualmente está activa en el Congo, los investigadores de la Complutense y la Universidad de Almería creen que el brote seguirá activo hasta principio de mayo 2020.

Sistema eficiente basado en superordenadores
“En los meses de agosto y septiembre, incluso octubre, la predicción que obtuvimos se mantuvo muy próxima a las cifras reales que ha dado la OMS. En noviembre, más o menos, empiezó a observarse algo más de diferencia entre las predicciones y la realidad, parece que sobre-estimamos el número de infectados y de fallecidos”, explica Míriam Ruiz Fernández, autora del modelo de optimización computacional aplicado a esta herramienta para la simulación de epidemias, y que desarrolló en el marco de su tesis doctoral, realizada en la Escuela Superior de Ingeniería almeriense.
A pesar de ese desajuste observado en los últimos meses, el modelo es totalmente fiable, ya que esa diferencia responde a que en septiembre comenzó a administrarse una nueva vacuna, además de la que ya estaba distribuyéndose, un aspecto que los especialistas de los grupos de Almería y Madrid no incorporaron a la simulación por producirse después.
“Nuestro modelo tiene en cuenta la eficiencia de las medidas de control que aplica el país o las autoridades sanitarias para tratar de frenar la epidemia. Por tanto, como se ha producido este cambio en las medidas de control, tenemos que volver a ajustar el modelo lanzando de nuevo nuestro algoritmo”, aclara la autora del modelo de optimización de esta herramienta de supercomputación.
El sistema emplea una media hora en hacer el cálculo para la simulación del comportamiento del brote de ébola, un tiempo computacional relativamente corto, ya que la epidemia afecta solamente a un país. Muy diferentes fueron las simulaciones realizadas del brote de ébola de 2014. En este caso, el sistema tardaba una media de 33 horas en realizar los cálculos necesarios, y todo ello con las técnicas de computación de altas prestaciones del clúster de la Universidad de Almería.
La cantidad de información manejada era inmensamente mayor, ya que en la simulación se tenía un espacio temporal de dos años y el cálculo se hacía para 176 países, para los que también se tenían en cuenta los movimientos migratorios y se priorizaba el ajuste de Guinea, Liberia y Sierra Leona, que fueron los países más afectados.
Optimiza un sistema multiobjetivo
La optimización del sistema de altas prestaciones descrita en la tesis de Míriam Ruiz Ferrández se ha basado en una nueva metodología multi-objetivo, adaptada a la herramienta Be-CoDiS, con la que se hacen las simulaciones de la propagación de la enfermedad tanto dentro de los países como entre ellos.
De esta manera se ha podido trabajar con un conjunto de valores para los parámetros epidemiológicos, “de modo que las predicciones se ajusten a los datos reales, tanto en la magnitud de la epidemia como en el momento crítico en que ésta despunta en cada país. Esto supone una gran mejora con respecto a las metodologías ya existentes, que calibraban algunos parámetros de Be-CoDiS mediante técnicas de regresión”, dice Ruiz Ferrández en su tesis.















